A fenda labial não sindrômica, com ou sem fenda palatina, é um defeito congênito comum em todo o mundo, devido a uma combinação de fatores ambientais e genéticos. O genome‐wide association studies relatou que o polimorfismo rs7078160 do Vax1 está intimamente relacionado à fenda labial não sindrômica, com ou sem fenda palatina em populações europeias. Estudos subsequentes mostraram os mesmos resultados nas populações mongol, japonesa, filipina e vietnamita etc. No entanto, pesquisas conflitantes foram relatadas na população chinesa.
ObjetivoInvestigar a associação entre o polimorfismo rs7078160 e fenda labial não sindrômica, com ou sem fenda palatina, em pacientes do sul da China.
MétodoTentamos investigar a distribuição do polimorfismo rs7078160 em 100 trios completos de pacientes (39 pacientes com fenda labial e palatina não sindrômica; 36 pacientes com fenda labial somente, não sindrômica; 25 com fenda palatina somente, não sindrômica e seus pais), da etnia Han do sul da China, e em 60 trios saudáveis selecionados como controle. Reação de polimerase em cadeia e o sequenciamento de Sanger foram uszados para genotipar o polimorfismo rs7078160 do Vax1 e tanto os casos‐controle quanto as associações baseadas na família foram analisadas.
ResultadosAs análises de caso‐controle revelaram que o polimorfismo rs7078160 estava significativamente associado a fenda labial não sindrômica, com ou sem fenda palatina (p=0,04) e fenda labial e palatina não sindrômica (p=0,01), mas não estava associado a pacientes com fenda labial somente não sindrômica e fenda palatina somente não sindrômica. A composição do genótipo de rs7078160 compreende AA homozigoto mutado, AG heterozigoto e GG homozigoto selvagem. Casos com genótipos AG+AA comparados com GG homozigotos mostraram um risco aumentado de fenda labial não sindrômica, com ou sem fenda palatina (p=0,04, OR=2,05, IC de 95%: 1,01−4,16) e fenda labial e palatina não sindrômica (p=0,01, OR=3,94, IC 95%: 1,34‐11,54). Além disso, não detectamos desequilíbrio de transmissão em rs7078160 (p=0,68).
ConclusãoEste estudo sugere que o polimorfismo rs7078160 foi um fator de risco para fenda labial não sindrômica, com ou sem fenda palatina, e o gene Vax1 está fortemente associado com fenda labial não sindrômica, com ou sem fenda palatina em populações da etnia Han do sul da China.
A fenda labial não sindrômica com ou sem fenda palatina (FLNS/P) é uma das malformações congênitas mais comuns em todo o mundo, com uma incidência de 1,2 em 1.000 nascidos vivos.1 Na China, a prevalência de FLNS/P é maior do que em outras populações étnicas (1,48 a 3,27 em 1.000 nascidos vivos).2,3 Atualmente, com o desenvolvimento da cirurgia plástica, mais pacientes com fenda labial ou palatina podem ser submetidos ao reparo cirúrgico;4 no entanto, eles ainda podem apresentar uma série de problemas, como deformação craniofacial e de fala. Além disso, embora instituições de caridade internacionais como a Smile Train tenham fornecido suporte para muitos pacientes, o tratamento da FLNS/P pode representar um custo econômico pesado para algumas famílias, especialmente nos países em desenvolvimento. Portanto, esclarecer as etiologias da FLNS/P é muito importante para o diagnóstico e a prevenção da doença.
As etiologias e a patogênese da FLNS/P são complexas, incluem fatores genéticos e ambientais ou uma combinação dos mesmos.3 Nos últimos anos, com o desenvolvimento de alta tecnologia de sequenciamento e metodologia estatística, o estudo de associação ampla do genoma (GWAS) foi feito para identificar genes candidatos envolvidos no desenvolvimento de FLNS/P.5 As evidências do GWAS identificaram dezenas de loci e genes causadores potenciais de FLNS/P, como o Irf6, Mafb, Grhl3, Abca4, Nog, Spry2, Tpm1, 8q21.3, Crebbp e Vax1.6–10 Entretanto, nas subsequentes reproduções, poucos desses loci/genes mostraram correlações consistentemente positivas em todos os estudos. Na verdade, para mais genes candidatos, resultados inconsistentes foram relatados em populações de diferentes etnias e regiões. Vax1 é um desses genes.
Um GWAS conduzido por Mangold et al. identificou um novo loci, rs7078160, associado a FLNS/P em 10q25 na população europeia.11 Posteriormente, Beaty et al. conduziram outro estudo GWAS em grupos europeus e asiáticos e seus achados confirmaram que o polimorfismo rs7078160 do Vax1 estava correlacionado com FLNS/P.6 Subsequentemente, estudos feitos em populações de mongóis, japoneses, filipinos e vietnamitas apoiaram os resultados acima.12–14 No entanto, pesquisas conflitantes foram relatadas na população chinesa e um estudo de Li et al. descobriu que rs7078160 do Vax1 não estava associado a FLNS/P na população da etnia Han do norte da China.15 Neste estudo, primeiramente investigamos a correlação entre os polimorfismos do Vax1 rs7078160 e o risco de FLNS/P na população de etnia Han do sul da China. Considerando que as amostras de trio completo podem fornecer dados mais confiáveis do que as amostras de caso‐controle, o desenho de caso/controle‐pais foi usado neste estudo, para que pudéssemos detectar as distribuições genotípicas e alélicas do polimorfismo de nucleotídeo único (SNP, do inglês Single Nucleotide Polymorphism) alvo enquanto analisamos os efeitos de origem parental.
MétodoAmostraO estudo foi conduzido no departamento de cirurgia oral e maxilofacial afiliado ao Stomatological Hospital of Zunyi Medical University (Zunyi, China) de 2016 a 2019. Cem pacientes FLNS/P (39 pacientes com fenda labial e palatina não sindrômica, FLPNS; 36 pacientes com fenda labial não sindrômica, FLNS; e 25 com fenda palatina não sindrômica, FPNS) e seus pais foram recrutados para serem avaliados no estudo. Sessenta trios saudáveis pareados foram selecionados como controle. O protocolo do estudo foi aprovado pelo comitê de ética em pesquisa da Zunyi Medical University (ZMU‐HG1/Set 25/16 data: 01/11/2016). O termo de consentimento livre e informado foi obtido dos pais ou outros responsáveis legais.
A avaliação clínica foi conduzida por cirurgiões orais e maxilofaciais experientes, para excluir os pacientes com FL/P sindrômica associados a qualquer outra deficiência congênita. Todos os casos de FLPNS, FLNS e FPNS eram unilaterais e nenhum dos pacientes tinha parentes afetados; pacientes com histórico familiar de doença genética também foram excluídos. Voluntários saudáveis sem histórico familiar de fenda labial e/ou palatina ou outra anormalidade foram escolhidos como controles. Todos os casos e controles eram da população chinesa de etnia Han, com a mesma distribuição de idade e gênero, e viviam no sul da China havia pelo menos três gerações. Três ml de sangue venoso periférico foram coletados de cada indivíduo e armazenados a ‐80°C.
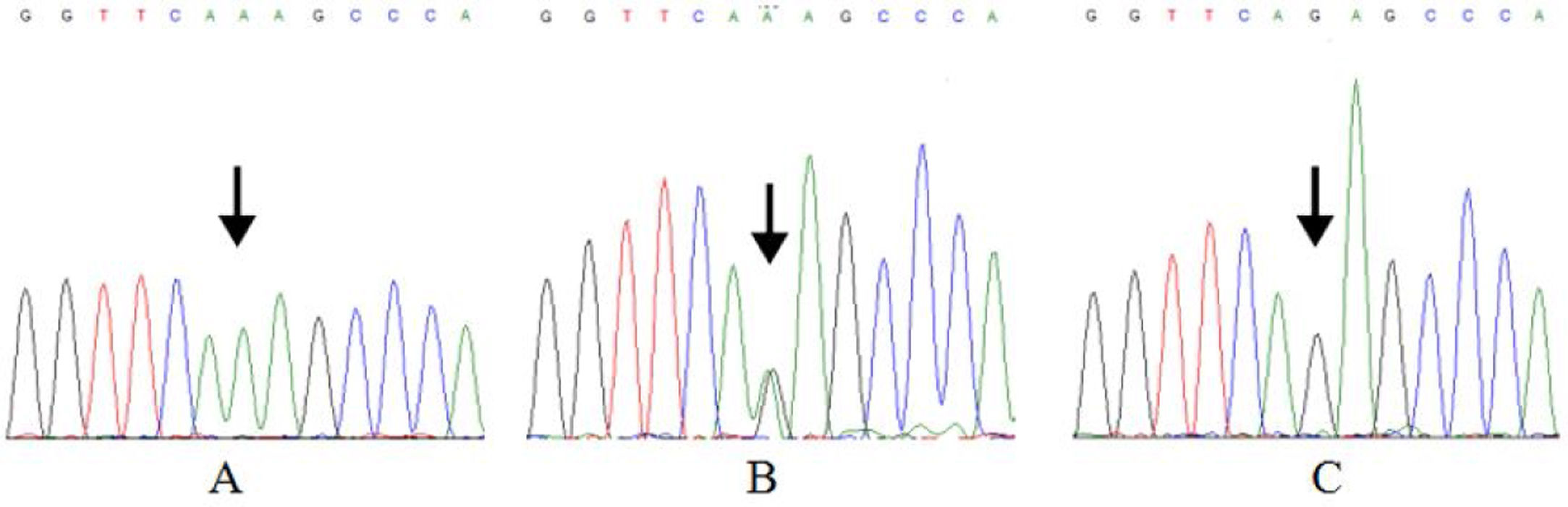
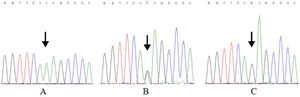
Extração de DNA, amplificação e sequenciamento por PCRO DNA genômico foi extraído da amostra de sangue com o kit TIANamp Blood DNA (Tiangen Biotech, Pequim, China). Os primers específicos de rs7078160 foram projetados de acordo com as sequências de referência do Vax1 no NCBI (National Center for Biotechnology Information, Bethesda, MD, EUA) pelo software Primer 5.0 (Premier Biosoft International, Palo Alto, CA, EUA). A sequência do primer forward foi 5’ TGGGAAGTGGGTGAGATGGA 3’, enquanto o primer reverso foi 5’ ATTGGGCGGACCCAGTAAAG 3’. Todas as amplificações de PCR foram feitas em um termociclador Gene Amp PCR system 9700 (Applied Biosystems, Foster City, CA, EUA). A mistura de reação consistiu em 10−30 ng de DNA genômico, 1μL de cada primer (10μmoL L‐1), 4μL de dNTPs (2,5 mmoL L‐1 de cada nucleobase), 5μL 10×de Tampão (com MgCl2) e 1,0U de Taq polimerase (TaKaRa, Dalian, China), adicionando‐se ddH2O a um volume de 50μL. A sequência de amplificação por PCR foi: 1) Desnaturação (94°C, 4min); 2) 34 ciclos incluindo desnaturação (94°C, 30s), annealing (58°C, 30s), extensão (72°C, 40s), 3) e Extensão final (72°C, 5min). Os produtos de PCR foram detectados por eletroforese em gel de agarose e purificados. O sequenciamento foi feito pelo BGI (Beijing Genomics Institute, Pequim, China). Alinhamos e editamos sequências com o software de sequenciamento de DNA Chromas 2.6.6 (Technelysium, Queensland, Austrália) e MEGA X.16 Os resultados do sequenciamento de DNA de rs7078160 são mostrados na figura 1.
Métodos estatísticosO frequências de genótipo e alélicas foram resumidos nos casos (grupo total, FLPNS e subgrupos FLNS e FPNS) e nos controles. O equilíbrio de Hardy‐Weinberg (EHW) afirma que ambas as frequências de alelos e genótipos em uma população de acasalamento aleatório permanecem constants. Com o intuito de demonstrar a estabilidade genética de nossos indivíduos, o EHW foi avaliado de acordo com a frequência do genótipo. Os resultados foram testados pelo teste qui‐quadrado com o software SPSS 16.0 (SPSS Inc., Chicago, IL, EUA). Em trios de casos‐pais, o teste de desequilíbrio de transmissão (TDT) foi conduzido com o pacote de softwareStatistical Analysis for Genetic Epidemiology (SAGE). O TDT é um teste baseado em família que oferece uma maneira poderosa de testar a transmissão de alelos‐alvo de pais heterozigotos para os filhos afetados. A odds ratio (OR) e intervalos de confiança de 95% (IC 95%) também foram calculadas e OR>1 significou um risco aumentado de FLNS/P. Todos os dados foram testados por valor de p bilateral e valores de p<0,05 foram aceitos como significativos.
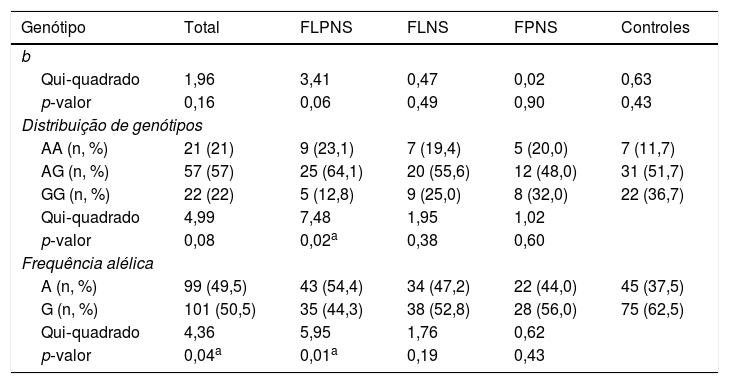
ResultadosA análise do equilíbrio de Hardy‐Weinberg revelou que todos os indivíduos apresentavam equilíbrio genético em rs7078160 (tabela 1). A distribuição dos genótipos e alelos da variante rs7078160 em ambos os grupos de casos e controles é apresentada na tabela 1. As proporções dos genótipos foram 21% AA, 57% AG, 22% GG nos casos e 11,7% AA, 51,7% AG, 36,7% GG em controles. A frequência do alelo G foi de 50,5% nos casos e 62,5% nos controles. As frequências alélicas de rs7078160 no grupo total de casos (p=0,04) e subgrupo FLPNS (p=0,01) foram substancialmente diferentes quando comparadas com o grupo controle; entretanto, as frequências alélicas nos subgrupos FLNS (p=0,19) e FPNS (p=0,43) não foram significantes quando comparadas ao grupo controle (tabela 1).
Distribuições genotípicas e alélicas do rs7078160 em pacientes com FLNS/P e controles (ap<0,05)
| Genótipo | Total | FLPNS | FLNS | FPNS | Controles |
|---|---|---|---|---|---|
| b | |||||
| Qui‐quadrado | 1,96 | 3,41 | 0,47 | 0,02 | 0,63 |
| p‐valor | 0,16 | 0,06 | 0,49 | 0,90 | 0,43 |
| Distribuição de genótipos | |||||
| AA (n, %) | 21 (21) | 9 (23,1) | 7 (19,4) | 5 (20,0) | 7 (11,7) |
| AG (n, %) | 57 (57) | 25 (64,1) | 20 (55,6) | 12 (48,0) | 31 (51,7) |
| GG (n, %) | 22 (22) | 5 (12,8) | 9 (25,0) | 8 (32,0) | 22 (36,7) |
| Qui‐quadrado | 4,99 | 7,48 | 1,95 | 1,02 | |
| p‐valor | 0,08 | 0,02a | 0,38 | 0,60 | |
| Frequência alélica | |||||
| A (n, %) | 99 (49,5) | 43 (54,4) | 34 (47,2) | 22 (44,0) | 45 (37,5) |
| G (n, %) | 101 (50,5) | 35 (44,3) | 38 (52,8) | 28 (56,0) | 75 (62,5) |
| Qui‐quadrado | 4,36 | 5,95 | 1,76 | 0,62 | |
| p‐valor | 0,04a | 0,01a | 0,19 | 0,43 | |
FLNS/P, fenda labial e palatina não sindrômica; FLPNS, Fenda labial e palatina não sindrômica; FLNS, Fenda labial não sindrômica; FPNS, Fenda palatina não sindrômica; EWH, Equilíbrio de Hardy‐Weinberg.
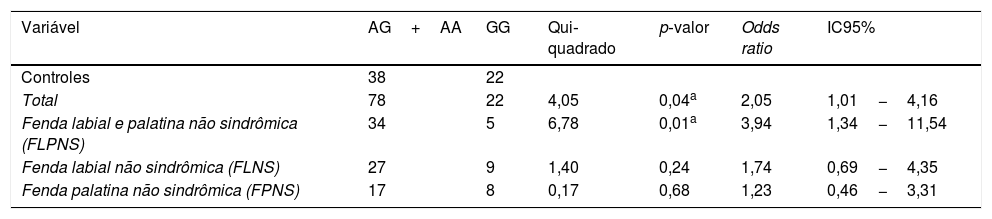
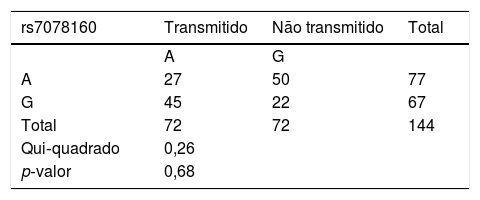
O OR e IC 95% entre AG+AA e GG para rs7078160 também mostraram diferenças significantes no grupo de casos totais (p=0,04, OR=2,05, IC 95%: 1,01‐4,16) e o subgrupo FLPNS (p=0,01, OR=3,94; IC 95%: 1,34−11,54), enquanto os resultados não foram significantes nos subgrupos FLNS (p=0,24) e FPNS (p=0,68) (tabela 2). O teste de desequilíbrio de transmissão foi feito para trios com estrutura genética em heterozigose e nenhum desequilíbrio transmitido estatisticamente significante foi detectado em rs7078160 (p=0,68) (tabela 3).
Resultados dos testes de associação com rs7078160 na fenda labial não sindrômica com ou sem fenda palatina (FLNS/P) (ap<0,05)
| Variável | AG+AA | GG | Qui‐quadrado | p‐valor | Odds ratio | IC95% |
|---|---|---|---|---|---|---|
| Controles | 38 | 22 | ||||
| Total | 78 | 22 | 4,05 | 0,04a | 2,05 | 1,01−4,16 |
| Fenda labial e palatina não sindrômica (FLPNS) | 34 | 5 | 6,78 | 0,01a | 3,94 | 1,34−11,54 |
| Fenda labial não sindrômica (FLNS) | 27 | 9 | 1,40 | 0,24 | 1,74 | 0,69−4,35 |
| Fenda palatina não sindrômica (FPNS) | 17 | 8 | 0,17 | 0,68 | 1,23 | 0,46−3,31 |
O importante papel etiológico de rs7078160 foi relatado pela primeira vez por Mangold et al. em um estudo de associação ampla do genoma em populações europeias com FLNS/P.11 O nucleotídeo de polimorfismo único (SNP) mais suscetível à FLNS/P em 10q25 foi rs7078160 no gene Vax1 (Ventral Anterior Homeobox 1). O Vax1 mostrou ser um gene crucial para o desenvolvimento do prosencéfalo basal e sistema visual em experimentos com animais e também participa da formação rostral de vertebrados.17 Slavotinek et al. relataram que a mutação em Vax1 está associada à fenda labial e palatina humana, ausência da glândula pineal, agenesia do corpo caloso, malformações no hipocampo e pequenos nervos ópticos.18
Até o momento, vários estudos confirmaram a associação entre rs7078160 e FLNS/P em diferentes populações étnicas, inclusive japoneses, poloneses, africanos da África Central e populações do Sudeste da Ásia.12‐14 Entretanto, resultados inconsistentes foram encontrados em chineses, quenianos e outros casos poloneses.15,19,20 Nos chineses, a pesquisa de Li et al. não identificou o risco de Vax1 em FLNS/P na população do norte da China.15 No entanto, recentemente um estudo conduzido por Zhang et al. descobriu que o gene Vax1 era um fator de risco para FLNS/P na população chinesa ocidental de etnia Han. 5 Além da diferença geográfica, os diferentes métodos dos estudos podem ser uma explicação. Os primeiros estudos usaram o desenho de caso‐controle, enquanto Zhang et al. adotaram o desenho de trio caso‐pais.5 Neste estudo, escolhemos a população de etnia Han do sul da China como amostra. As frequências alélicas de rs7078160 no grupo FLPNS foram substancialmente diferentes quando comparadas ao grupo controle (p=0,02, respectivamente) (tabela 1). O OR e IC de 95% entre AG+AA e GG mostraram diferença significativa no grupo de casos totais (p=0,04, OR=2,05, IC de 95%: 1,01‐4,16) e subgrupo FLPNS (p=0,01, OR=3,94, 95% IC: 1,34‐11,54), sugeriu que o alelo A de rs7078160 pode ser um fator de risco para FLNS/P, mas não está associado a FLNS e FPNS (tabela 2). O teste TDT não detectou desequilíbrio transmitido significante em rs7078160 (p=0,68) (tabela 3). Esse resultado foi conflitante com os estudos anteriores de FLNS/P de Li et al., já que eles não encontraram associação de Vax1 e FLNS/P na população de etnia Han do norte da China.15 A inconsistência pode ser devido à diferença geográfica e ao desenho de amostragem diferente; o trio pais‐caso completo pode fornecer informações genéticas mais abrangentes em comparação com o desenho de caso‐controle.
De maneira coletiva, este estudo demonstrou que o polimorfismo rs7078160 está envolvido no desenvolvimento de FLNS/P em indivíduos da etnia Han do sul da China. Nosso estudo forneceu outra evidência para a etiologia de Vax1 em FLNS/P da população chinesa de etnia Han. Entretanto, nosso estudo ainda tem várias limitações potenciais. Primeiro, nos concentramos em apenas um SNP, o rs7078160; o estudo de mais SNPs candidatos poderia aumentar a significância de nosso estudo. Em segundo lugar, devido às dificuldades de coletar as amostras do trio completo, o número de casos e trios de controle era relativamente limitado. O aumento do número de indivíduos fortaleceria nossos resultados. Em terceiro lugar, as interações ambientais e os fatores maternos durante a gravidez não foram avaliados em nosso estudo. Mais esforços ainda são necessários para esclarecer a associação mais profunda entre hereditariedade e meio ambiente.
ConclusãoEste estudo revelou que o polimorfismo rs7078160 está envolvido no desenvolvimento de FLNS/P na população de etnia Han do sul da China, demonstra que Vax1 pode estar associado a FLNS/P. Esses achados podem ser úteis para ampliar nosso conhecimento sobre a etiologia da FLNS/P.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
A todos os indivíduos que tornaram este estudo possível, especialmente aos pacientes, seus pais e aos controles que colaboraram voluntariamente. Esta pesquisa recebeu apoio do Top Science and Technology Talent Project in Higher Education Institution of Guizhou Province (Qian‐Jiao‐He KY Zi [2016]080), Outstanding Young Talent Project of Zunyi Medical University (17zy‐002) e Science and Technology Project of Zunyi Science and Technology Bureau (ZSKHZC NS 2019‐15).
Como citar este artigo: Wang Q, Sun S, Song Q, Hu H, An J, Liu J. The risk of nonsyndromic cleft lip with or without cleft palate and Vax1 rs7078160 polymorphisms in southern Han Chinese. Braz J Otorhinolaryngol. 2021;87:718–22.
A revisão por pares é da responsabilidade da Associação Brasileira de Otorrinolaringologia e Cirurgia Cérvico‐Facial.