Salivary gland tumors (SGTs) are rare head and neck malignancies consisting of a spectrum of tumors with different biological behaviors.
ObjectiveIn this study we aimed to find out differential expression of microRNA profiles between benign and malignant SGTs.
MethodsWe investigated the possible role of 95 microRNAs in the 20 patients with salivary gland tumors with comparison of 17 patients without malignancy or salivary gland diseases. Sixteen of the tumors were benign (seven pleomorphic adenomas, nine Warthin tumors), four of them were malignant (two squamous cell carcinomas, one high grade mucoepidermoid carcinoma, one adenocarcinoma). Serum and saliva samples were collected from both patients and control group. Tissue samples of tumor masses were also collected from patient group.
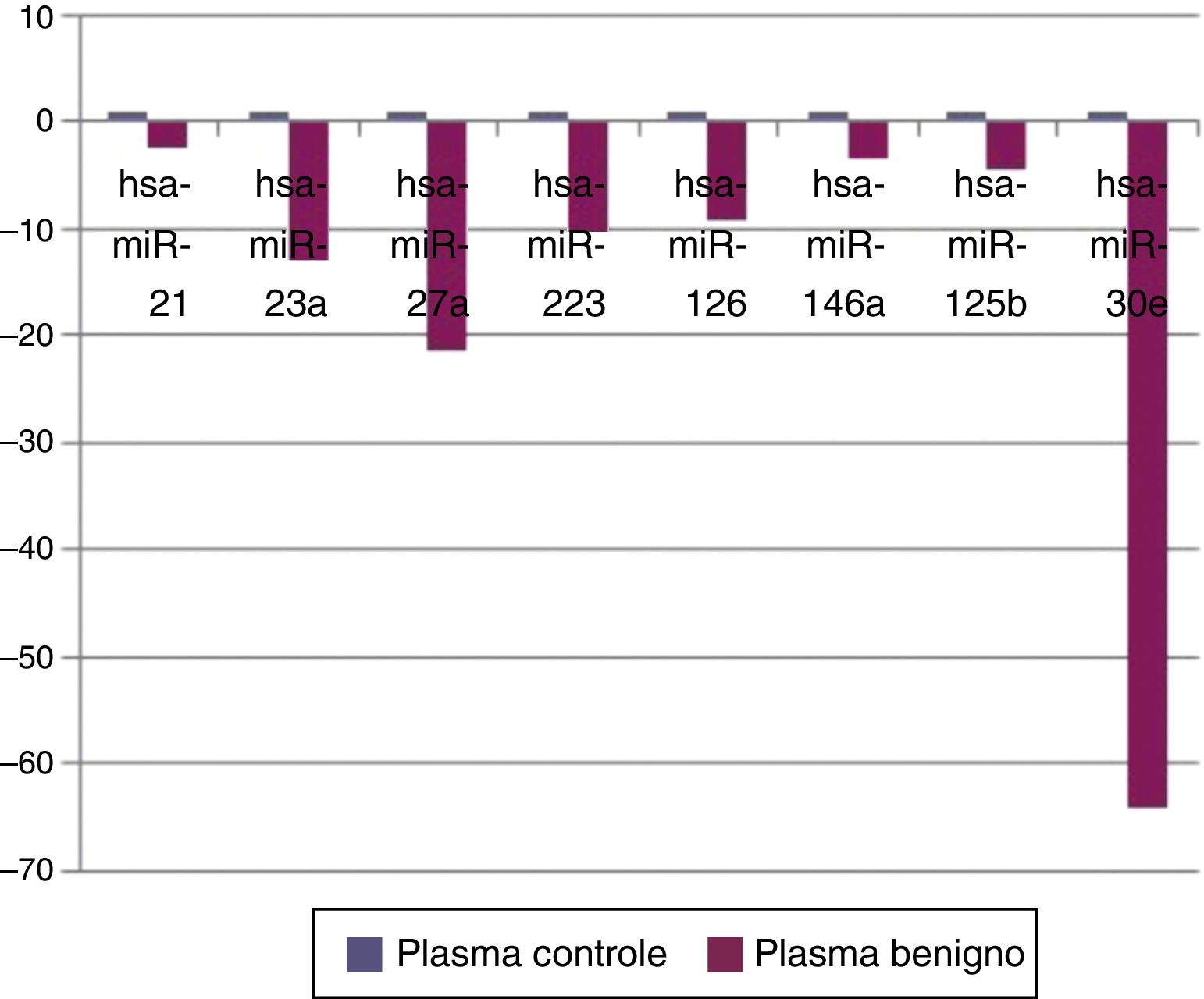
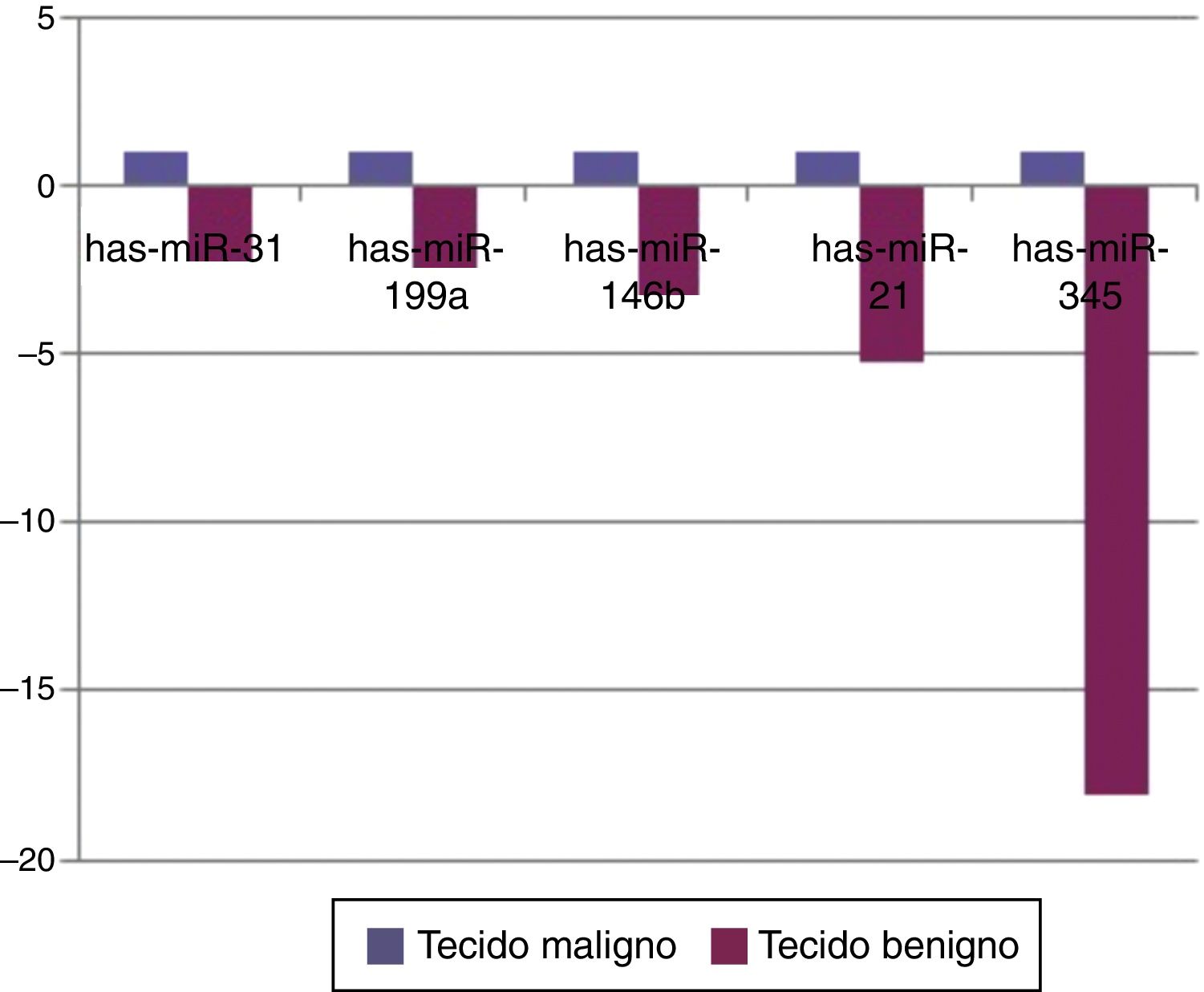
ResultsAmong studied microRNAs miR‐21, miR‐23a, miR‐27a, miR‐223, miR‐125b, miR‐126, miR‐146a, miR‐30e were down regulated in the benign group compared to control group in the serum samples (p‐values are 0.04, 0.00005, 0.00005, 0.0022, 0.031, 0.00008, 0.044, and 0.0007, respectively). When tissue samples were studied miR‐21, miR‐31, miR‐199a‐5p, miR‐146b, miR‐345 were up‐regulated in the malignant group compared to benign group (p values are 0.006, 0.02, 0.013, 0.013, 0.041, respectively). miR‐30e showed statistically significant up‐regulation in malignant tumor group's plasma samples compared to benign group (p=0.034). There was no statistically significant difference in saliva samples between groups.
ConclusionOur results showed that different microRNAs may play role in salivary tumor pathogenesis according to biological behavior. Although there was no difference in saliva samples between groups, according to tissue and serum samples miR‐21 and 30e may have an important role; since they were down‐regulated in benign tumors whereas up‐regulated in malignant ones.
Os tumores da glândula salivar (TGS) são lesões malignas raras de cabeça e pescoço que consistem em um espectro de tumores com diferentes comportamentos biológicos.
ObjetivoNeste estudo, tivemos como objetivo identificar a expressão diferencial de perfis de microRNA entre TGS benignos e malignos.
MétodoInvestigamos a possível participação de 95 microRNA em 20 pacientes com tumores de glândulas salivares comparados com 17 pacientes sem doença maligna ou doenças das glândulas salivares; 16 dos tumores eram benignos (sete adenomas pleomórficos, nove tumores de Warthin), quatro eram malignos (dois carcinomas espinocelulares, carcinoma mucoepidermoide de alto grau, um adenocarcinoma). As amostras de soro e saliva foram coletadas de pacientes e do grupo controle. Amostras de tecido dos tumores também foram colhidas do grupo de pacientes com tumores.
ResultadosEntre os microRNA estudados, miR‐21, miR‐23a, miR‐27a, miR‐223, miR‐125b, miR‐126, miR‐146a, miR‐30e foram infrarregulados no grupo benigno em comparação com o grupo controle nas amostras do soro (os valores de p são 0,04, 0,00005, 0,00005, 0,0022, 0,031, 0,00008, 0,044 e 0,0007, respectivamente). Quando as amostras de tecido foram estudadas, miR‐21, o miR‐31, o miR‐199‐5p, miR‐146b, o miR‐345 foram suprarregulados no grupo maligno em relação ao grupo benigno (valores de p são 0,006, 0,02, 0,013, 0,013, 0,041, respectivamente). O miR‐30e apresentou suprarregulação estatisticamente significativa em amostras de plasma do grupo de tumor maligno em relação ao grupo benigno (p=0,034). Não houve diferença estatisticamente significativa em amostras de saliva entre os grupos.
ConclusãoNossos resultados mostraram que diferentes microRNA podem desempenhar um papel na patogenia do tumor salivar de acordo com o comportamento biológico. Embora não tenha havido diferença em amostras de saliva entre os grupos, de acordo com as amostras de tecido e de soro, miR‐21 e 30e podem ter um papel importante, já que foram infrarregulados nos tumores benignos enquanto suprarregulados nos tumores malignos.
Os tumores de glândulas salivares (TGS) compreendem apenas 3 a 5% de todos os tumores malignos de cabeça e pescoço; eles têm pelo menos 24 tipos diferentes de acordo com a classificação de 2005da Organização Mundial da Saúde.1 Entre eles, o adenoma pleomórfico é o tumor benigno mais comum, enquanto o carcinoma mucoepidermoide é o maligno mais comum.2 A patogenia exata e o caminho para a transformação maligna de tumores de glândulas salivares não são bem conhecidos. Embora o tabagismo e o consumo de álcool sejam fatores de risco importantes para carcinomas espinocelulares (CEC), de cabeça e pescoço, eles não são admissíveis para tumores de glândulas salivares e exposições ocupacionais não parecem participar da patogenia.3
Os microRNA (miRNA) são um grupo de 21‐25 RNA endógenos não codificantes de nucleotídeos que têm como alvo a codificação do gene no nível pós‐transcricional.4,5 Eles estão envolvidos em vários processos biológicos importantes, tais como desenvolvimento, diferenciação, proliferação e apoptose.5 Eles podem se comportar como oncogenes ou genes supressores de tumor; sua suprarregulamentação ou infrarregulação pode participar da carcinogênese.6 Entre os cânceres de cabeça e pescoço, os CEC e, entre os tumores malignos da glândula salivar, os carcinomas adenoides císticos (CAC) são os cânceres mais comuns estudados para um possível papel dos miRNAs na patogenia da lesão maligna.4–7 Neste estudo, investigamos o possível papel dos miRNA na patogenia do tumor da glândula salivar. Até onde sabemos, este é o primeiro estudo a comparar os níveis de miRNA séricos, da saliva e de tecidos entre as neoplasias de glândulas salivares.
MétodoA aprovação do comitê de ética local foi obtida para o nosso estudo. Foram incluídos 20 pacientes com tumores de glândulas salivares e 17 controles saudáveis pareados para sexo e idade sem qualquer distúrbio de glândula salivar ou doenças sistêmicas. A média de idade para o tumor da glândula salivar foi de 53,1 anos e a média de idade para o grupo controle foi de 46,4 anos. Havia dez pacientes do sexo masculino (50%) e dez do feminino (50%) no grupo de tumores e nove pacientes do sexo masculino (52,9%) e oito do feminino (47,1%) no grupo controle. Não houve diferença estatisticamente significativa em relação à idade (p=0,251) e sexo (p=0,858) entre os grupos de tumores e de controle. Do grupo de tumor, havia 16 tumores benignos e quatro malignos. Os malignos eram dois CEC, um carcinoma mucoepidermoide de alto grau e um adenocarcinoma. Apenas os carcinomas espinocelulares primários foram levados para o estudo de acordo com critérios de inclusão de Gaughan;8,9 os carcinomas espinocelulares metastáticos foram excluídos. Os benignos eram nove tumores de Warthin e sete adenomas pleomórficos. A lista dos tumores se encontra resumida na tabela 1. No grupo do tumor, um (25%) de quatro no grupo maligno e cinco (31,25%) de 16 no grupo benigno eram do sexo feminino. Em relação a diferenças de sexo e idade entre os grupos de tumores malignos e benignos, não houve diferença estatística para sexo (p=0,807) e idade (p=0,9355).
Amostras de sangue de 5ml foram coletadas para 7,5% EDTA com tubos de grupo de pacientes no pré‐operatório e no grupo controle. Após centrifugação, os soros foram colhidos e armazenados a ‐80°C.
Coleta de salivaApós a cateterização dos ductos de glândula salivar envolvidos, com limão como sialogogo, 200μL de saliva da glândula envolvida foram coletados. A saliva foi misturada com 200μL de solução de RNAlater (Qiagen Inc., Valência, CA). Após duas horas em temperatura ambiente, elas foram armazenadas a ‐80°C em congelador.10 Também foram coletados 200μlda saliva a partir do grupo controle.
Coleta de tecidoAs amostras de tecido foram tomadas apenas do grupo de tumores da glândula salivar. Durante a cirurgia do tumor da glândula salivar, uma amostra de tecido tumoral de 3‐4 mm3 foi usada para análise. As amostras de tecido foram colocadas em 1mL de solução de RNAlater (Qiagen Inc., Valência, CA). Após duas horas em temperatura ambiente, foram armazenadas a ‐80°C em congelador. Os tecidos foram seccionados com uma lâmina cirúrgica e homogeneizados com pilão e almofariz. O isolamento do microRNA a partir de tecido homogeneizado foi feito com kit de isolamento de microRNA (Roche Diagnostics GmbH, Mannheim, Alemanha), de acordo com as instruções do fabricante.
Perfil de expressão de microRNAApós os microRNA serem isolados a partir do soro, da saliva e do tecido por um kit de isolamento de microRNA (Roche Diagnostics, GmbH, Mannheim, Alemanha), de acordo com as instruções do fabricante, as amostras de RNA foram convertidas em DNAc com o uso do kit de miScript II RT (Qiagen); as amostras de DNAc foram pré‐amplificadas, com kit miScript microfluidics Pre‐AMP (Qiagen); e a análise de qRT‐PCR foi feita com ensaios de miScript miRNA (Qiagen) com arranjo dinâmico 96,96 (Fluidigm) no BioMark System (Fluidigm). Basicamente, foram estudados 95 tipos de microRNA.
Análise de dadosOs resultados qRT‐PCR foram analisados com o método 2‐ΔΔCt.11,12 Resumidamente, a suprarregulação, ou infrarregulação de microRNA, foi comparada entre os grupos. O teste t de Student foi usado para analisar as diferenças de idade e o teste do qui‐quadrado foi usado para analisar as diferenças de sexo entre os grupos. Os dados de expressão foram controlados para distribuição normal com o teste de Shapiro‐Wilk. De acordo com os resultados, os dados não foram distribuídos normalmente. O teste U de Mann‐Whitney foi usado para detectar diferenças de expressão de miRNA em amostras de soro, tecidos e saliva. O valor p<0,05 foi considerado estatisticamente significativo.
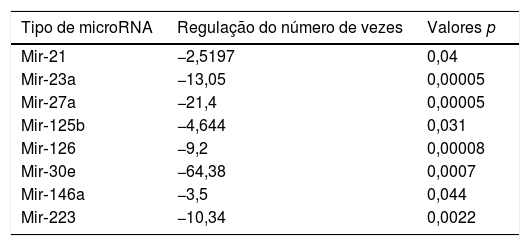
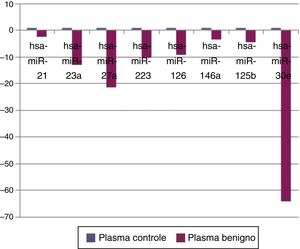
ResultadosQuando os tumores benignos foram comparados com o grupo controle, oito miRNA (miR‐21, miR‐23a, miR‐27a, miR‐223, miR‐125b, miR‐126, miR‐146a, e miR‐30e), como mostrado na figura 1, tinham infrarregulação estatisticamente significativa no grupo de tumores benignos em amostras de soro. A tabela 2 resume a regulação do número de vezes e valores p desses miRNA. Não houve suprarregulação estatisticamente significante desses miRNA em amostras de soro.
MicroRNA mostra mudanças estatisticamente significativas por comparação entre amostras de plasma de tumor benigno e grupos controle com valores p
| Tipo de microRNA | Regulação do número de vezes | Valores p |
|---|---|---|
| Mir‐21 | −2,5197 | 0,04 |
| Mir‐23a | −13,05 | 0,00005 |
| Mir‐27a | −21,4 | 0,00005 |
| Mir‐125b | −4,644 | 0,031 |
| Mir‐126 | −9,2 | 0,00008 |
| Mir‐30e | −64,38 | 0,0007 |
| Mir‐146a | −3,5 | 0,044 |
| Mir‐223 | −10,34 | 0,0022 |
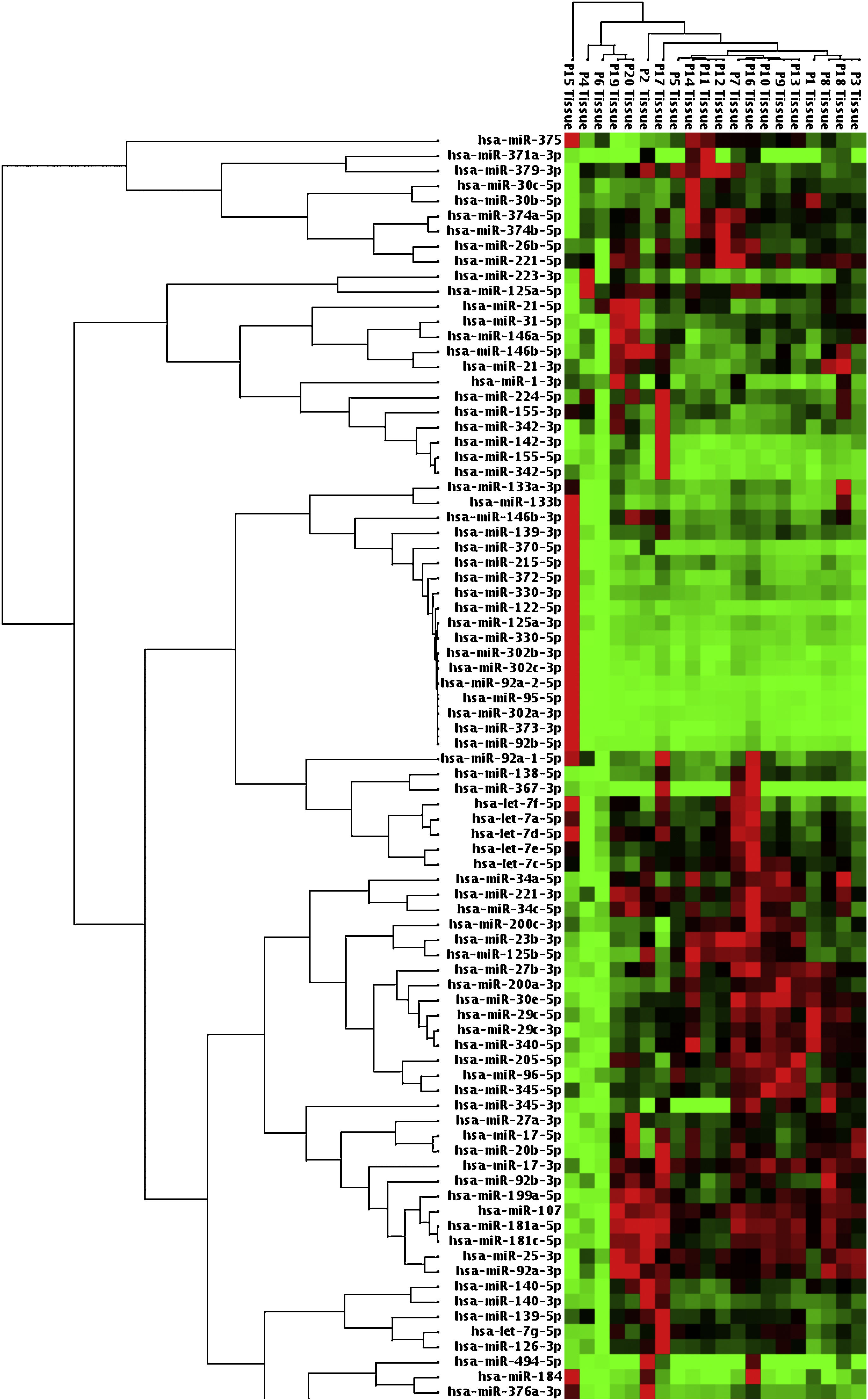
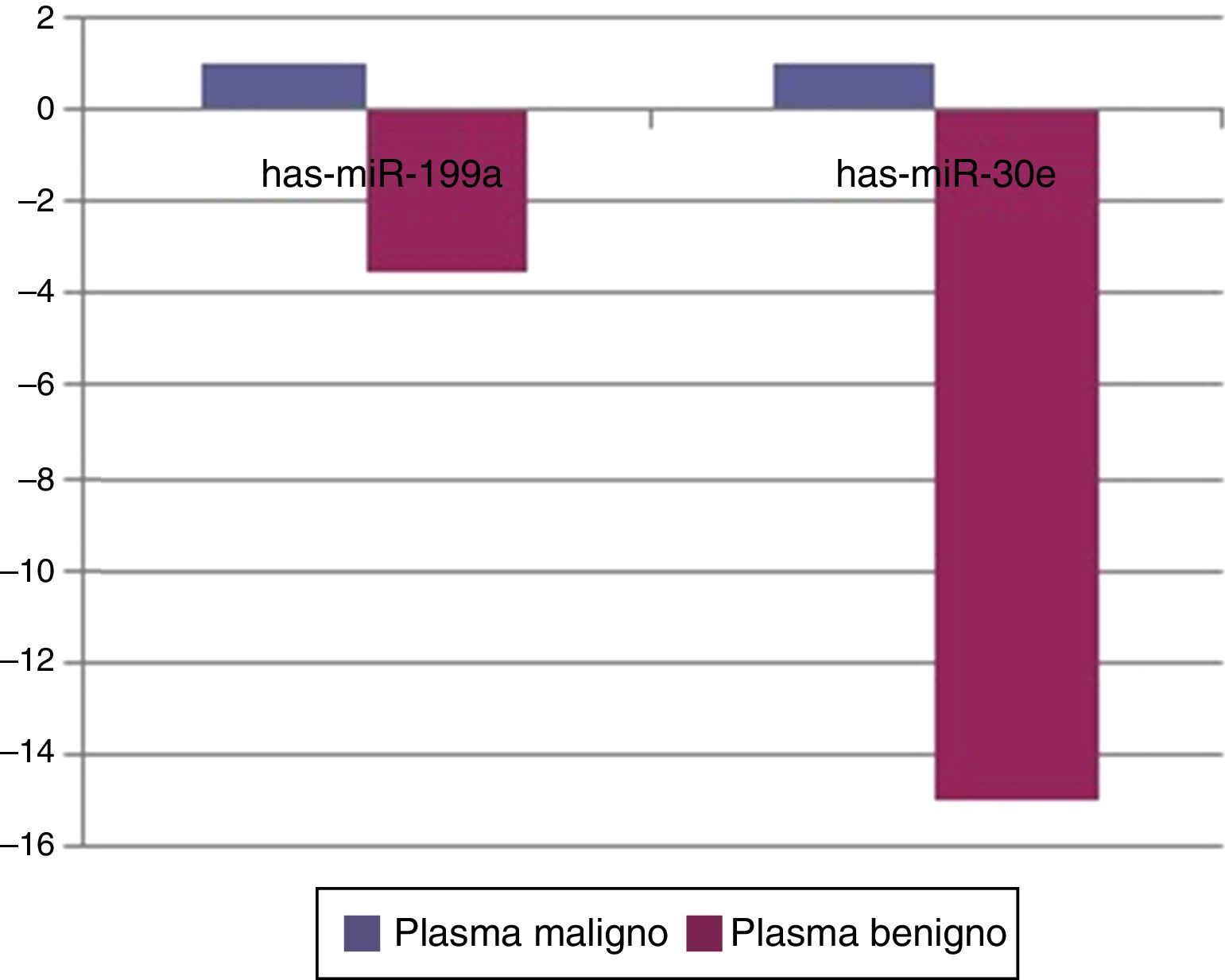
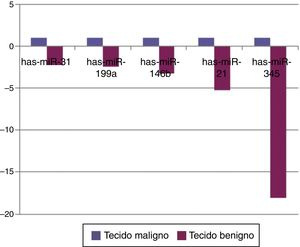
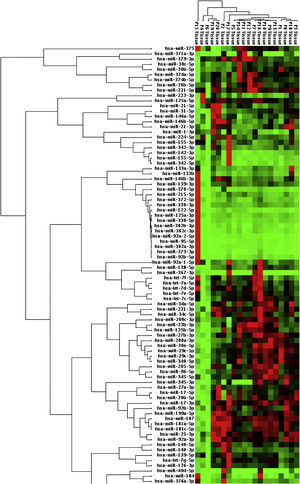
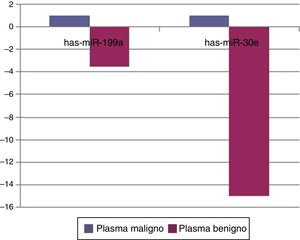
Quando os pacientes do grupo de tumores malignos foram comparados com os do grupo de tumores benignos em amostras de tecido, cinco miRNAs (miR‐21, 31, 199A, 146B, 345) apresentaram suprarregulação estatisticamente significativa no grupo do tumor maligno (fig. 2). A tabela 3 resume os valores de regulação do número de vezes no grupo de tumores malignos com valores de p. O diagrama do heat map de agrupamento de miRNA em amostras de tecido foi mostrado na figura 3. Entre esses miRNA, miR‐199 também mostrou suprarregulação estatisticamente significativa (p=0,042) em amostras de soro do grupo maligno, em comparação com o grupo benigno. O miR‐30e apresentou suprarregulação de 15,06 vezes (p=0,034) em amostras de plasma do grupo de tumor maligno, em comparação com o grupo benigno (fig. 4).
Expressão de microRNA de grupo de tumor maligno comparado com o grupo de tumor benigno em amostras de tecido que mostra mudanças estatisticamente significativas
| Tipo de MicroRNA | Regulação do número de vezes | Valores p |
|---|---|---|
| Mir‐21 | +5,23 | 0,0006 |
| Mir‐31 | +2,24 | 0,02 |
| Mir‐199a | +2,36 | 0,013 |
| Mir‐146b | +3,21 | 0,013 |
| Mir‐345 | +18,063 | 0,041 |
Quando o grupo de tumor maligno foi comparado com o grupo controle, miR‐23a apresentou suprarregulação de 10,5 vezes (p=0,02) em amostras de soro de tumores malignos (dados não mostrados).
Não houve diferença estatisticamente significativa em amostras de saliva entre os grupos benigno, maligno e controle.
DiscussãoDesde que foram encontrados pela primeira vez no nematódeo Caenorhabditis elegans, em 1993,13 miRNAs têm sido investigados por seus prováveis papéis em estados fisiológicos normais e doentes em vários estudos. Eles são uma classe de pequenos RNA endógenos que têm como alvo a expressão do gene no nível pós‐transcricional e que regulam negativamente a tradução do RNA mensageiro.4,14 Após transcrito via RNA‐polimerase II ou III, eles têm vários comportamentos biológicos importantes, tais como proliferação, diferenciação, apoptose e motilidade.4,14 Essas características importantes os tornam cruciais no desenvolvimento fisiológico dos tecidos e as anomalias na sua transcrição podem levar a diferenciação aberrante e carcinogênese.6
Para o desenvolvimento embrionário normal das glândulas salivares, os papéis de miRNAs têm sido investigados pelos autores. Em um modelo murino, Jevnaker e Osmundsen15 descobriram que havia miRNA específicos de tecidos na glândula submandibular para o desenvolvimento normal, como o miR‐28, 150, 222, 299, 322, 329, 341, 375 e 429. Os miRNAs miR‐23, 27‐a, 223, 125b, 126, 30e, que foram expostos em nosso estudo como infrarregulados no grupo de tumor benigno, também foram expressos em glândula submandibular de embriões de camundongos. Além disso, alguns membros do agrupamento de miRNA 17‐92, como miR‐19b, miR‐20a e miR‐92, foram altamente expressos durante o desenvolvimento da glândula submandibular de camundongos.14 O miR‐21, um dos miRNA mais importantes estudados em nosso relato, é também um miRNA crucial para o desenvolvimento embriológico da glândula submandibular e sua morfogênese de ramificação. Demonstrou‐se que ele se expressa no tecido mesenquimal da glândula submandibular de camundongos; esta expressão foi suprarregulada pelo fator de crescimento epidérmico, que tem papel fundamental na ramificação do ducto da glândula submandibular.16
Os papéis do desenvolvimento fisiológico quase normal dos miRNA na carcinogênese também são investigados. Como os primeiros sinais de seu provável papel na carcinogênese foram encontrados por Calin et al.,17 em 2002, pela infrarregulação de miR‐15a, miR‐16‐1 na leucemia linfoide crônica, vários estudos sobre a relação entre miRNA e carcinogênese foram publicados. Entre os cânceres de cabeça e pescoço, o carcinoma espinocelular é amplamente estudado por provável papel dos miRNA. Embora exista uma grande diferença entre os estudos, miR‐21 é o único miRNA constante que apresenta suprarregulação nos cânceres espinocelulares de cabeça e pescoço.6 Ele tem efeitos antiapoptóticos, de proliferação celular, com promoção de invasão celular e metástase; então, a suprarregulação desse miRNA também pressupõe mau prognóstico em cânceres de cabeça e pescoço.18 Em nosso estudo, houve uma suprarregulação cinco vezes maior (p=0,0006) de miR‐21 em amostras de tecidos tumorais malignos de glândulas salivares, em comparação com tumores benignos. Houve também uma infrarregulação de 2,51 vezes (p=0,047) em amostras plasmáticas de grupo benigno, em comparação com o grupo controle. Esse miRNA pode ter um papel crucial na patogenia do tumor de glândula salivar e no grupo benigno.
Em nosso estudo, demonstrou‐se que miR‐125b é suprarregulado em amostras de soro de tumores benignos de glândulas salivares. O miR‐125b e miR‐100 são dois miRNAs importantes que mapeiam o cromossomo 11. As alterações nesse cromossomo podem levar a cânceres orais e carcinoma mucoepidermoide de glândulas salivares.19,20 No estudo de Hui et al.,18 a expressão de miR‐125b também foi infrarregulada no carcinoma espinocelular de cabeça e pescoço, assim como em nosso estudo. A infrarregulação desse miRNA pode causar aumento da proliferação celular e carcinogênese.19
O miR‐23a e miR‐27a pertencem ao agrupamento de miR 23a∼27a∼24‐2 localizado no cromossomo 9q22. Eles têm múltiplas funções em ambos os estados saudável e de doença, tais como ciclo celular, proliferação, diferenciação e hipertrofia cardíaca21 A literatura que investiga o papel desses miRNA na carcinogênese é conflitante. Os miR‐23a e miR‐27a mostraram ser infrarregulados no CEC oral,22 enquanto, recentemente, Peng et al. apontaram que o miR‐23a promove quimiorresistência à quimioterapia com cisplatina no CEC de língua.23 Os miR‐23a e 27‐a são infrarregulados na leucemia promielocítica aguda e suprarregulados na leucemia mieloide aguda, leucemia linfoblástica aguda, no câncer gástrico e câncer hepático.21 Considerando a literatura recente, é evidente que esses miRNA podem comportar‐se de maneira diferente na patogenia de diferentes cânceres; eles podem atuar como gene supressor de tumor em um câncer e como oncogene em outro. Em nosso estudo, demonstrou‐se que miR‐23a e 27a eram infrarregulados em amostras de soro de tumores de glândulas salivares benignos. O miR‐23a também foi infrarregulado no grupo de tumor maligno, em comparação com o grupo controle em amostras de soro. O miR‐23a pode comportar‐se como um gene supressor de tumor em ambos os tumores da glândula salivar: benignos e malignos.
Quando amostras de tecido e plasma foram comparadas entre os tumores, houve uma suprarregulação de 2,3656 vezes (p=0,01357) de miR‐199‐5p em amostras de tecido e suprarregulação de 3,5 vezes (p=0,042) em amostras de soro de tumores malignos. Também foi demonstrado que esse miARN é suprarregulado no câncer de cólon, hepatocelular, gástrico e no melanoma maligno.24,25 No estudo de Liu et al.,26 o miR‐31 foi suprarregulado no CEC oral e os níveis diminuíram após o tratamento em relação ao possível papel desse miRNA como um marcador nesse câncer. Em nosso estudo, houve uma suprarregulação de 2.244 vezes (p=0,0208) desse miRNA no grupo de tumor salivar maligno em amostras de tecido. O miR‐146b mostrou‐se importante fator prognóstico para pacientes com câncer de tireoide papilar, maior expressão desse miRNA em células tumorais diminuiu a sobrevida.27 A expressão de miR‐146b também foi aumentada em células de câncer de tireoide anaplásicas.28 No entanto, para câncer de mama, a expressão de miR‐146b causou uma redução da capacidade metastática, suprimiu a atividade de NF‐κB.29 Em nosso estudo, houve uma suprarregulação de 3,21 vezes (p=0,013) de miR‐146b no grupo de tumor maligno, em comparação com o grupo benigno, nas amostras de tecido tumoral. Descobriu‐se que o miR‐345 tem um valor prognóstico no câncer de próstata30 e colorretal.31 Em nosso estudo, foi suprarregulado 18 vezes (p=0,041) no grupo maligno. Para o miR‐30e, demonstrou‐se que foi suprarregulado no câncer hepatocelular32 e infrarregulado no câncer de tireoide anaplásico.33 Em nosso estudo, o miR‐30e apresentou suprarregulação de 15,06 (p=0,034) em amostras de plasma do grupo de tumor maligno, em comparação com o grupo benigno, enquanto houve uma infrarregulação no grupo de tumor benigno em relação ao grupo controle (p=0,0007) em amostras de plasma.
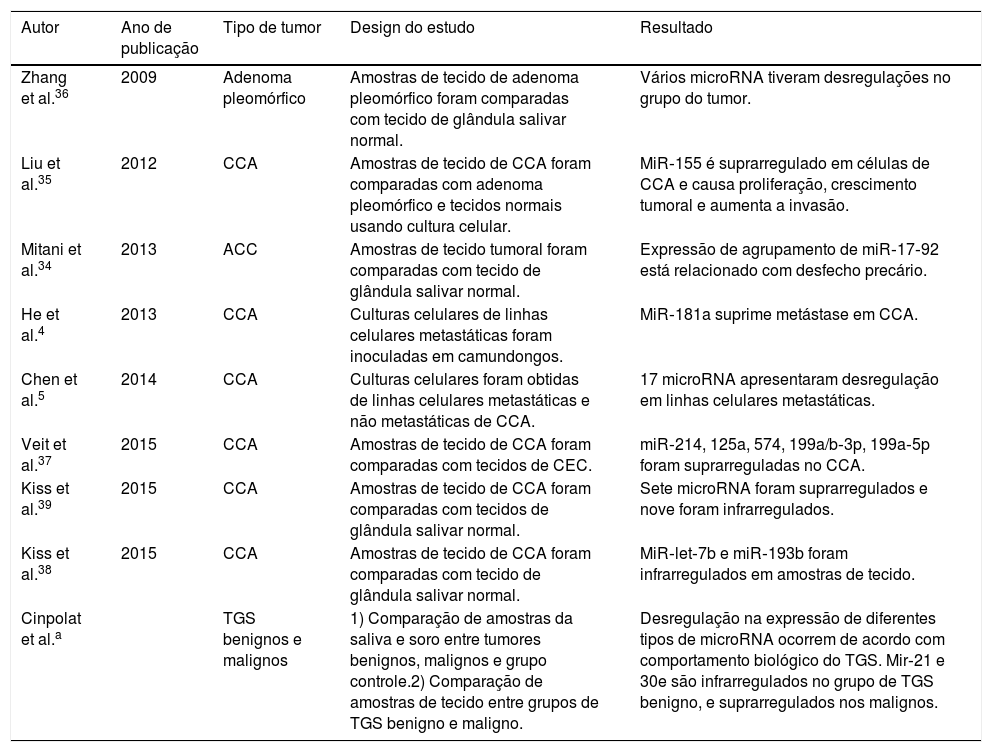
Estudos sobre a relação entre tumores de glândulas salivares e miRNAs são limitados. Mitani et al. estudaram os perfis de miRNA de amostras de CCA em comparação com tecidos salivares normais. Eles observaram que a superexpressão de miR‐17 e miR‐20a foi relacionada com desfecho precário.34 He et al. demonstraram que a expressão de miR‐181a em células tumorais de CCA diminuiu o potencial metastático do tumor.4 Liu et al. especularam que a supressão de miR‐155 pode inibir a proliferação celular e o crescimento do tumor para CCA.35 Chen et al. observaram que 17 miRNA diferentes tiveram mudanças estatisticamente significativas em fold‐change de linhas celulares no CCA metastático, em comparação com os não metastáticos.5 Entre os tumores de glândulas salivares benignos, os miRNAs foram investigados para o possível papel na patogenia dos adenomas pleomórficos. Zhang et al. relataram que 17 miRNA diferentes foram suprarregulados em amostras de tumores, inclusive os miR‐21.36 Esse resultado não foi consistente com o nosso estudo. Nele, houve uma infrarregulação de miR‐21 em amostras de soro de pacientes do grupo de tumores benignos, em comparação com o grupo controle. Essa diferença poderia ter duas explicações. Primeiramente, nosso grupo de tumor benigno era composto por múltiplas doenças, e não só de adenoma pleomórfico. Em segundo lugar, as amostras de soro foram estudadas no grupo controle, e não nas biópsias de tecido. Em outro estudo, Veit et al. compararam os perfis de miRNA de CCA e CEC da região de cabeça e pescoço. Os miR‐214, 125a, 574, 199a/b‐3p e 199a‐5p foram suprarregulados no grupo de CCA.37 Em nosso estudo, o miR‐199 também foi suprarregulado em amostras de tecido do grupo maligno. Recentemente, Kiss et al. 38 demonstraram possível papel da infrarregulação de miR‐let‐7b e miR‐193b na patogenia do CCA da glândula salivar. Novamente, em outro estudo, Kiss et al. 39 encontraram sete miRNA superexpressos e nove miRNA subexpressos em amostras de tecido salivar de CCA. Estudos publicados anteriormente, com a comparação do nosso, foram resumidos na tabela 4.
Estudos previamente publicados relativos à relação entre microRNA e tumores de glândula salivar
| Autor | Ano de publicação | Tipo de tumor | Design do estudo | Resultado |
|---|---|---|---|---|
| Zhang et al.36 | 2009 | Adenoma pleomórfico | Amostras de tecido de adenoma pleomórfico foram comparadas com tecido de glândula salivar normal. | Vários microRNA tiveram desregulações no grupo do tumor. |
| Liu et al.35 | 2012 | CCA | Amostras de tecido de CCA foram comparadas com adenoma pleomórfico e tecidos normais usando cultura celular. | MiR‐155 é suprarregulado em células de CCA e causa proliferação, crescimento tumoral e aumenta a invasão. |
| Mitani et al.34 | 2013 | ACC | Amostras de tecido tumoral foram comparadas com tecido de glândula salivar normal. | Expressão de agrupamento de miR‐17‐92 está relacionado com desfecho precário. |
| He et al.4 | 2013 | CCA | Culturas celulares de linhas celulares metastáticas foram inoculadas em camundongos. | MiR‐181a suprime metástase em CCA. |
| Chen et al.5 | 2014 | CCA | Culturas celulares foram obtidas de linhas celulares metastáticas e não metastáticas de CCA. | 17 microRNA apresentaram desregulação em linhas celulares metastáticas. |
| Veit et al.37 | 2015 | CCA | Amostras de tecido de CCA foram comparadas com tecidos de CEC. | miR‐214, 125a, 574, 199a/b‐3p, 199a‐5p foram suprarreguladas no CCA. |
| Kiss et al.39 | 2015 | CCA | Amostras de tecido de CCA foram comparadas com tecidos de glândula salivar normal. | Sete microRNA foram suprarregulados e nove foram infrarregulados. |
| Kiss et al.38 | 2015 | CCA | Amostras de tecido de CCA foram comparadas com tecido de glândula salivar normal. | MiR‐let‐7b e miR‐193b foram infrarregulados em amostras de tecido. |
| Cinpolat et al.a | TGS benignos e malignos | 1) Comparação de amostras da saliva e soro entre tumores benignos, malignos e grupo controle.2) Comparação de amostras de tecido entre grupos de TGS benigno e maligno. | Desregulação na expressão de diferentes tipos de microRNA ocorrem de acordo com comportamento biológico do TGS. Mir‐21 e 30e são infrarregulados no grupo de TGS benigno, e suprarregulados nos malignos. |
CCA, carcinoma adenoide cístico; miR, microRNA; TGS, tumores de glândula salivar.
Em nosso estudo, descobrimos que miR‐21 e miR‐30e foram suprarregulados no grupo de TGS malignos e infrarregulados no grupo benigno. Quando esses miRNA são investigados quanto aos seus genes‐alvo, RPS7 e LIMCH1 são os genes‐alvo comuns desses miRNA. (Esses genes foram pesquisados com sistemas de banco de dados de microRNA Mirwalk e Mirtarbase).40,41 O gene RPS7 encontrado no cromossomo 2 codifica uma proteína ribossômica pertencente à família de S7E e o gene LIMCH1 encontrado no cromossomo 4 codifica proteínas de ligação ao íon de zinco.40,41 Em estudos futuros, as proteínas de ligação de íons de zinco e as proteínas ribossômicas pertencentes à família de S7E podem ser novas moléculas‐alvo para diferenças no comportamento biológico dos tumores das glândulas salivares em amostras de soro e/ou resultados de biópsia aspirativa com agulha fina.
Embora o tamanho da nossa amostra de tumores malignos pareça pequena, houve artigos publicados que investigaram as funções de miRNA na carcinogênese de TGS com tamanho de amostra semelhante. No estudo de Veit et al.,37 foram comparadas cinco amostras de tecido de tumor de CCA com dez amostras de tecido tumoral de CEC. No estudo de Liu et al.,35 houve comparação entre dez casos de CCA e quatro casos de adenoma pleomórfico e oito tecidos da glândula parótida normais. Recentemente, Kiss et al. 39 publicaram seu estudo com apenas dois casos de CCA salivares. Consideramos nossos resultados significativos para a investigação da patogenia de miRNA em TGS.
ConclusãoComo resultado e sob a luz de artigos previamente publicados e do estudo atual, pode‐se especular que o miRNA desempenhe um papel na patogenia do tumor de glândula salivar. A desregulação do tipo de miRNA difere de acordo com o comportamento biológico. O miR‐21 e o miR‐30E podem ter um papel crítico no desenvolvimento do tumor de glândula salivar com os genes‐alvo RPS7 e LIMCH1.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
O financiamento deste estudo foi mantido pela Unidade de Projetos de Pesquisa Acadêmica da Mersin University.
Como citar este artigo: Cinpolat O, Unal ZN, Ismi O, Gorur A, Unal M. Comparison of microRNA profiles between benign and malignant salivary gland tumors in tissue, blood and saliva samples: a prospective, case‐control study. Braz J Otorhinolaryngol. 2017;83:276–84.
A revisão por pares é da responsabilidade da Associação Brasileira de Otorrinolaringologia e Cirurgia Cérvico‐Facial.